NEBNext 酶学转化法甲基化建库试剂盒 收藏
Download:
- isoschizomers |
- compatible ends |
- single letter code
相关产品
NEBNext 酶学转化法甲基化建库相关产品
NEBNext 酶学转化法甲基化建库试剂盒
NEBNext 甲基化建库酶学转化法模块
NEBNext Q5U™ 预混液
NEBNext 酶学转化法甲基化建库多样本接头引物(双端检索引物)
概述
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq™) 提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
优势
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块
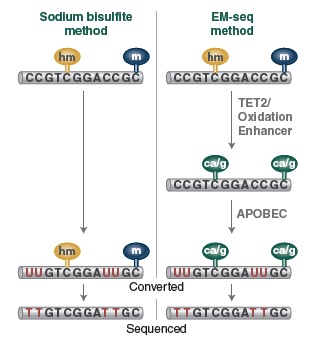
产品特点
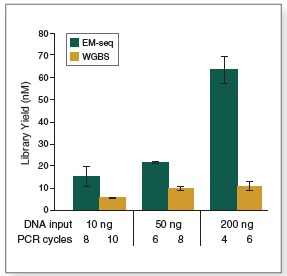
EM-Seq 能得到更高的文库产量。采用Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。
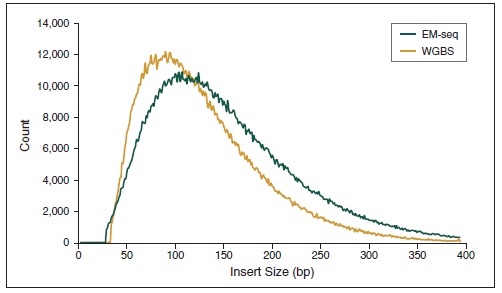
NEBNext 酶学转化法甲基化文库可获得更长的插入片段 。采用Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq (2 X 76 bases) 测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。
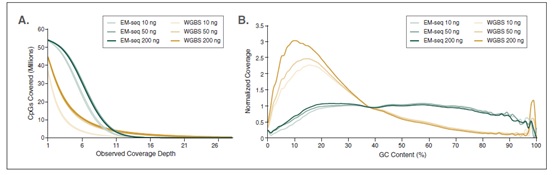
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点,并能实现更卓越的 GC 均一覆盖度。采用Covaris S2 仪器将 10 ng, 50 ng, 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000 (2 X 100 bases) 测序,使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。
A: CpG 位点检测:通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
B: GC 覆盖度:使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时 (0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。
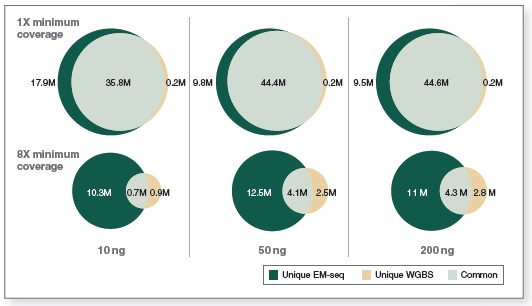
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点。采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。
